La transformation génétique du pneumocoque s’organise sur les fourches de réplication de son génome
La transformation génétique naturelle est un mécanisme de transfert horizontal d’ADN qui promeut le brassage de l’information génétique chez les bactéries par recombinaison homologue. Dans ce travail conduit chez Streptococcus pneumoniae (le pneumocoque), un pathogène opportuniste majeur de l’homme, les scientifiques ont visualisé en temps réel par microscopie à fluorescence l’initiation de l’intégration de l’ADN dans le génome lors de la transformation et ainsi révélé que la recombinaison de l’ADN s’amorce spécifiquement sur les fourches de réplication du génome. Ces résultats obtenus par des scientifiques du Laboratoire de microbiologie et génétique moléculaire1 (LMGM/CBI) sont publiés dans la revue PNAS.
© Calum Johnston
La recombinaison homologue (RH) est un mécanisme universel et essentiel pour la maintenance de l’intégrité des génomes et le brassage de l’information génétique. Elle se décline en un ensemble de voies, qui se distinguent par les effecteurs qui les composent. Toutes ces voies de RH procèdent par une série ordonnée d’échanges de brins d’ADN complémentaires, suivant un mécanisme commun catalysé par une recombinase conservée dans tous les organismes. Les étapes initiales de la RH conduisent à l’assemblage de cette enzyme en un polymère dynamique sur un des brins d’ADN échangés. Ainsi filamentée sur l’ADN simple-brin (ADNsb) et définissant le filament ‘pré-synaptique’ de la RH, la recombinase procède ensuite à la recherche d’une séquence complémentaire dans une molécule d’ADN double-brin (ADNdb) pour y catalyser leur appariement. La maturation de cette synapse d’ADN est propre à chaque voie de RH, tout comme les étapes initiales qui coordonnent la formation d’ADNsb et le recrutement de la recombinase sur ce brin d’ADN.
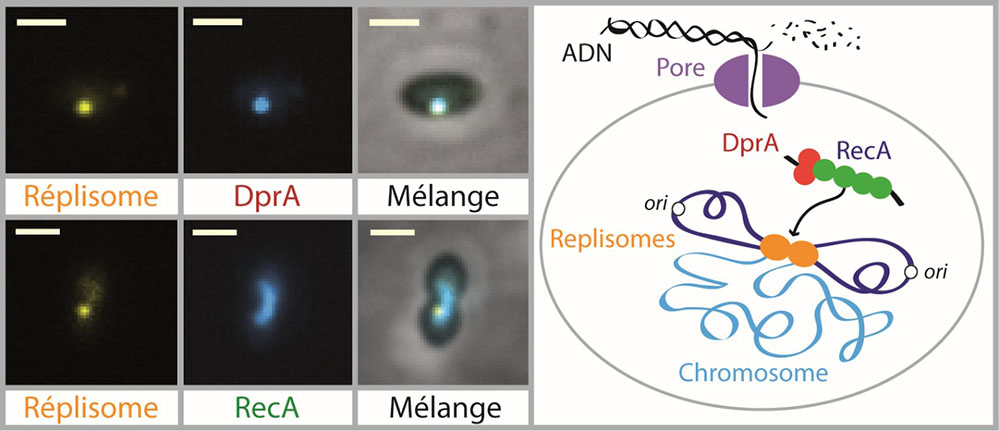
Chez les bactéries, la transformation génétique naturelle est une voie de RH dédiée au brassage de leur information génétique. Elle procède par la capture et l’internalisation dans les cellules d’ADNdb présent dans le milieu extérieur, suivi ensuite de son intégration à des sites d’homologie du génome. A l’étape de transition de son transport au travers de la membrane cytoplasmique, l’ADNdb exogène est maturé en fragments linéaires simple-brin, recombinés dans le génome par la recombinase RecA commune à toutes les voies de RH bactériennes. Un effecteur essentiel et spécifique de la transformation est la protéine DprA, démontrée génétiquement et biochimiquement interagir à la fois avec l’ADNsb et RecA pour médier la formation du filament pré-synaptique de la RH. De manière remarquable, il a été établi chez la bactérie Streptococcus pneumoniae (le pneumocoque) que la durée du trajet d’un fragment d’ADN radiomarqué parfaitement homologue à une région précise de son génome, depuis son ajout aux cellules jusqu’à son intégration, est de l’ordre de la minute.
Comment s’organise la formation des filaments pré-synaptiques de RH de la transformation, ainsi que l’accession et l’interaction de ces filaments avec le génome dans une si courte fenêtre de temps sont les questions adressées dans cette étude. En utilisant des fusions fonctionnelles de DprA ou RecA avec des protéines fluorescentes, les scientifiques ont visualisé en temps réel les étapes d’initiation de la RH de la transformation du pneumocoque. Cette étude spatiotemporelle de la RH de la transformation a révélé que la filamentation présynaptique de RecA sur l’ADNsb médiée par DprA prend appui sur les fourches de réplication du génome, ceci dans la fenêtre de temps de la minute après ajout d’ADN exogène aux cellules. A la lumière de ce résultat inédit, les scientifiques proposent qu’en se formant à partir des fourches de réplication les filaments pré-synaptiques de RH de la transformation peuvent accéder idéalement et balayer efficacement l’ADN du génome pour trouver puis s’apparier à une séquence d’ADN complémentaire.
- 1Tutelles : CNRS, UT3
Bibliographie
The RecA-directed recombination pathway of natural transformation initiates at chromosomal replication forks in Streptococcus pneumoniae.
Johnston CHG, Hope R, Soulet AL, Dewailly M, De Lemos D, Polard P.
PNAS, (2023) 120(8):e2213867120.
doi: 10.1073/pnas.2213867120.
Epub 2023 Feb 16.PMID: 36795748